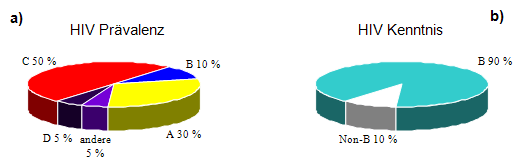|
HIV-1 Resistenz und TropismusDas primäre Ziel einer antiretroviralen Therapie besteht in einer maximalen Viruslastreduktion. Wird dieses Ziel nicht erreicht, kann bei medikamentösem Selektionsdruck das Virus gegen Bestandteile der Therapie resistent werden. 1. Grundlagen der ResistenzentwicklungDas Erbmaterial des HIV besteht aus je 2 RNA-(Ribonukleinsäure)-Strängen, die die genetischen Informationen des Virus beinhalten. Drei Nukleotide bilden ein sogenanntes Codon und codieren jeweils für eine Aminosäure der Proteinsequenz. Eine Veränderung der Nukleotidfolge eines Codons, eine Mutation, kann den Einbau einer anderen Aminosäure zur Konsequenz haben, was die Proteinfunktion beeinträchtigen kann. Position der Aminosäure auf dem Gen (hier die HIV-Protease) I84V Wildtyp-Aminosäure Aminosäure der Mutante Isoleucin (ATA) Valin (GTA) Eine Strukturveränderung der viralen Proteine durch untypische Aminosäuresequenzen kann dazu führen, dass antiretrovirale Wirkstoffe gar nicht oder nur noch teilweise wirksam sind. Man spricht von einem Wirkverlust der antiretroviralen Medikamente oder einer Resistenz. 2. Wie entstehen Resistenzen?Eine wichtige Rolle bei der Entstehung von Mutationen im HIV-Genom und der daraus resultierenden Resistenz spielt das viruseigene Enzym Reverse Transkriptase (RT). Es handelt sich dabei um ein Protein, welches die virale RNA in DNA umschreibt, die dann ins Zellgenom integriert wird. Hierbei werden relativ häufig andere Nukleotide als in der Originalsequenz vorgegeben eingebaut, wodurch Aminosäureaustausche in der letztendlich resultierenden Proteinsequenz entstehen können. Bedingt durch die hohe Vermehrungsrate des HI-Viruses (es werden in unbehandelten Patienten täglich ca. 10 Milliarden neue Viren gebildet) und der fehlerhaft arbeitenden RT, kann theoretisch jede Position des ca. 10.000 Nukleotide umfassenden HIV Genoms täglich ausgetauscht werden. Die ist ein Hauptgrund für die hohe Variabilität des HI-Virus. Es gibt unterschiedliche Arten von Mutationen:
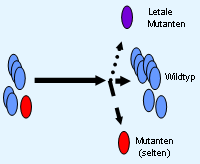
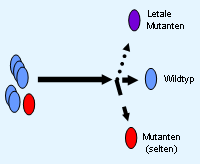
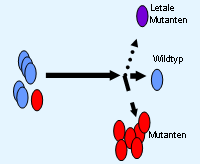
Die Entwicklung einer HIV-1 Resistenz im Individuum hängt von der Generierung und Selektion entsprechender resistenzrelevanter Mutationen ab.Hat ein Virus einmal eine oder mehrere resistenzassoziierte Mutationen erlangt, die ihm eine verminderte Empfindlichkeit gegen antiretrovirale Medikamente verleiht, so hat er gegenüber dem Virus-Urtyp (Wildtyp) einen Vermehrungsvorteil. Der Selektionsvorteil für das mutierte Virus resultiert in einer dominierenden Viruspopulation von resistenten Viren (Abb.1). Die Resistenzentwicklung verläuft oftmals nicht nach dem 'Alles-oder-Nichts-Prinzip', sondern stellt einen graduellen Prozeß dar. Bei vielen antiretroviralen Substanzen wird eine vollständig ausgeprägte Resistenz erst durch die Anhäufung von mehreren Mutationen bewirkt. Man verwendet daher neben den Einstufungen resistent und sensitiv noch ein bis zwei intermediär resistente Graduierungen. Abb.1: Auswirkung des medikamentösen Selektionsdrucks auf unterschiedliche Viruspopulationen. siehe auch Kapitel 5.2.: Quasispezies
Je effektiver die Virusreplikation durch antiretrovirale Medikamente unterdrückt wird, desto weniger Mutationen können durch Fehler der Reversen Transkriptase in das Virus-Genom integriert werden und desto länger bleibt die Wirksamkeit antiretroviraler Wirkstoffe bestehen. 3. Wie sind Resistenzen meßbar?Die Viruslast im Plasma kann durch molekularbiologische Methoden quantitativ bestimmt werden und ist einer der wichtigsten Laborparameter zur Kontrolle des Therapieerfolges. Das Therapieziel ist die Viruslast unter die Nachweisgrenze von 50 HIV-1 RNA Kopien pro Milliliter Plasma zu senken. Ein frühes Indiz einer sich etablierenden Resistenz ist ein Anstieg der Viruslast des unter antiretroviraler Therapie stehenden Patienten. Bevor eine Resistenzanalyse durchgeführt wird, sollte abgeklärt werden, ob die meßbare Viruslast nicht nur ein einmaliges Ereignis ist. Insbesondere niedrige Werte von bis zu wenigen Hundert Kopien pro Milliliter, auch 'Blip' genannt, sollten kontrolliert werden. Eine messbare Viruslast kann auch bedingt durch eine Medikamentenabsorbtionsstörung oder durch eine unzureichende Adhärenz seitens des Patienten verursacht sein. Zur Messung der viralen Resistenzentwicklung können zwei Methoden verwendet werden: die genotypische und die phänotypische Resistenzanalyse. 3.1 Genotypische ResistenzanalyseBei der genotypischen Resistenzanalyse werden Nukleotidsequenzbereiche des Protease-, des Reverse Transkriptase-, des Integrase- und des gp41-Gens molekularbiologisch analysiert. Die Methodik der genotypischen Resistenzanalyse läßt sich grob unterteilen in:
Im letzten Schritt lassen sich vorliegende Veränderungen (Mutationen) im Vergleich zu einer Wildtypsequenz unter anderem an resistenzrelevanten Stellen ausmachen. Anhand der Mutationen werden Vorhersagen über die Wirksamkeit von antiretroviralen Substanzen getroffen. Bei der Bestätigung einer Medikamentenresistenz muß anschließend entschieden werden, ob die bestehende Therapie auf andere Wirkstoffe und Medikamente umgestellt oder entsprechend ergänzt werden soll.
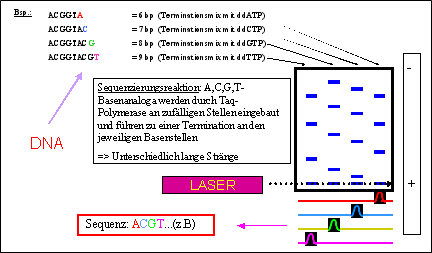
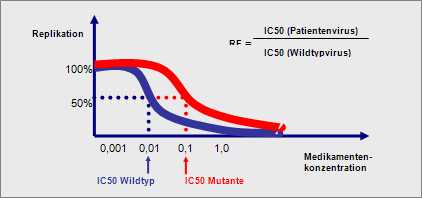
Abb.2: Vereinfachte Darstellung der Sequenzierungs-Technik Die Vorteile einer genotypischen Resistenzanalyse liegen in einer schnellen Verfügbarkeit der Ergebnisse. Des-weiteren können hier Mutationen identifiziert werden, die Aufschluss über eine in der Vergangenheit bestandene oder eine sich entwickelnde Resistenz geben können (Transitionsmutation). Einige Vor- und Nachteile der genotypischen- bzw. der phänotypischen Resistenzanalyse sind in Tab.1 aufgeführt. 3.2 Phänotypische ResistenzanalyseIm Rahmen der phänotypischen Resistenzanalyse wird das Maß der Empfindlichkeit des Virus gegenüber einer Medikamentensubstanz bestimmt. Dabei werden unterschiedliche Verdünnungsreihen antiretroviraler Wirkstoffe an einem rekombinierten HIV-Isolat, welches den zu analysierenden Genbereich des Patienten-Isolates molekularbiologisch integriert bekommen hat, getestet. Die durch Mutationen bedingte verminderte Empfindlichkeit des HIV auf bestimmte Substanzen wird üblicherweise als x-fache Erhöhung des IC50 (entspricht der Konzentration einer Substanz, die nötig ist um eine 50%ige Hemmung der Virusreplikation zu erzielen) im Vergleich zur Wildtyp-Referenz als phänotypischer Resistenzfaktor (RF) angegeben (s. Abb.3).
Abb.3: Darstellung des IC50 von Wildtyp und Mutante Ein wesentlicher Vorteil der phänotypischen Resistenzanalyse liegt durch die direktere Messung in der Beurteilung von Kreuzresistenzen und in der Abwägung der Resistenz von neuen antiretroviralen Substanzen. Zur Beurteilung der Resistenzeinstufung wurden hierbei unterschiedliche Schwellenwerte (Cut offs) entwickelt:
Der klinische Cut Off ist der wichtigste und aussagekräftigste Schwellenwert. Es werden meist ein oberer und ein unterer Cut-off definiert. Am unteren Cut-off ist das virologische Ansprechen bereits leicht vermindert, ab dem oberen Cut-off-Wert ist, wenn überhaupt, nur noch ein geringes virologisches Ansprechen zu erwarten. Für neuere Medikamente fehlt oftmals aus Datenmangel dieser Schwellenwert. Es ist zu berücksichtigen, dass die phänotypische Resistenzanalyse keine EBM-Leistung ist. Die Durchführung kann somit nur im Rahmen von Studien und Projekten oder nach vorheriger Antragsstellung und genehmigter Kostenübernahme seitens der Krankenkasse stattfinden.
Tab.1: Einige Vor- und Nachteile der genotypischen bzw. phänotypischen Resistenzanalyse
Zur Durchführung der Resistenzanalyse muss eine Mindestmenge an Viren vorhanden sein. Bei einer Viruslast von weniger als 200 Kopien/ml kann die Resistenzanalyse häufig nicht durchgeführt werden. 4. Interpretation von genotypischen ResistenzergebnissenAufgrund der Verfügbarkeit von zahlreichen antiretroviralen Substanzen und der Komplexität der möglichen Mutationsmuster wurden Interpretationshilfen (Algorithmen) entwickelt. Diese Interpretationssysteme erstellen auf Basis der variablen Gewichtung von vorliegenden Mutationen Vorhersagen über die verbleibende Wirksamkeit der antiretroviralen Substanzen. Es existieren derzeit zwei grundlegende Varianten zur Interpretation der Ergebnisse von genotypischen Resistenzanalysen, regelbasierte und bioinformatische Systeme. Regelbasierte Algorithmen beinhalten meist in vitro und in vivo Daten basierend auf Studienveröffentlichungen und Expertenwissen. Zur Zeit sind diverse Interpretations-Systeme aus diesem Bereich im Internet frei zugänglich. Einige von ihnen ermöglichen die direkte Analyse der DNA-Sequenz und/oder die Möglichkeit manuell Mutationen einzutragen. Als Beispiel seien nachfolgende frei verfügbare Interpretationshilfen genannt:
Bioinformatische Systeme hingegen stützen sich auf mathematische Modelle, die auf Geno- und Phänotyp Korrelationen basieren. Da der phänotypische Resistenzfaktor berechnet/ermittelt wird, spricht man hier vom virtuellen Phänotyp. Als Beispiel sei hier Geno2Pheno http://www.geno2pheno.org/ und der kommerziell erhältliche Virtual Phenotype der Firma Virco genannt. 5. Andere Faktoren, die einen Einfluß auf die Resistenzentwicklung haben5.1 PrimärresistenzVon Primärresistenz spricht man, wenn eine Person mit einem bereits resistenten Virus infiziert wird. Die Wahrscheinlichkeit mit einem primär resistenten Stamm infiziert zu werden liegt in Deutschland bei 10%. Diesbezüglich ist es mehr als empfehlenswert vor dem Start der ersten Therapie eine Resistenzanalyse durchzuführen, damit sichergestellt wird, dass nur aktive Substanzen eingesetzt werden. 5.2 Vortherapie und QuasispeziesMit und ohne antiretroviraler Therapie entstehen täglich unterschiedliche Virusvarianten. Somit existiert im Organismus nicht nur eine einheitliche Viruspopulation, sondern es entstehen viele unterschiedliche Viren (Quasispezies), deren Anzahl im Laufe des Infektionszeitraums zunimmt. Im Blut sind meist nur die Virusvarianten mit den höchsten Wachstumsraten nachweisbar. Die Detektionsgrenze liegt mit der weitverbreiteten Sequenzierungstechnik bei 20%. Wenn medikamentöser Selektionsdruck gesetzt wird, können sich resistente Viren entwickeln oder schon existierende resistente Viren aus dem zellulären Reservoir, in dem sie in Form von proviraler DNA integriert sind, erneut heranwachsen. Somit sollte ein Resistenzbefund immer unter Berücksichtigung der Vortherapie bewertet werden. 5.3 HIV non- B SubtypenWährend der Ausbreitung von HIV, haben sich weltweit zahlreiche Subtypen entwickelt, die sich teilweise stark voneinander unterscheiden können (s. Kapitel HIV-Subtypen). Antiretrovirale Substanzen, die heute zum Einsatz kommen, wurden im nordamerikanischen und europäischen Raum entwickelt und am hier prädominanten HIV-1 Subtyps verifiziert. Daher sind auch die meisten vorliegenden Daten zur Resistenzentwicklung aus den Beobachtungen mit diesem Subtyp generiert worden. Jedoch macht dieser Subtyp in der Populationsdynamik nur 10% der globalen HIV-Pandemie aus (s. Abb. 4). In einigen europäischen Ländern mit hohem Migrationsanteil in der Bevölkerung, wie zum Beispiel in Belgien und Portugal, ist der Anteil an non-B Subtypen schon größer als 40%. In Deutschland je nach Region bis zu 25 %. Globales Paradox
Abb. 4: Globale Prävalenz des HIV-Subtyps B mit entsprechendem Anteil an derzeitigen Kenntnissen über HIV Es ist bekannt, dass unter Umständen das gleiche Medikament bei verschieden HIV-Subtypen unterschiedliche Mutationen hervorrufen kann. Manche dieser Mutationswege können eine breitere Kreuzresistenz bewirken. Des weiteren können durch genetische Prädisposition bei einigen HIV-non B Subtypen Mutationen leichter entstehen. Dieses sollte nach neuesten Erkenntnissen auch bei der Resistenzinterpretation berücksichtigt werden. Diesbezüglich sollte bei der Resistenzanalyse routinemäßig der HIV Subtyp mit ermittelt werden. Das ist via Internet anhand der Reversen Transkriptase und Protease Sequenz für den weit überwiegenden Teil der Sequenzen schnell und kostenfrei möglich. 5.4 Kreuzresistenz, kompensatorische Mutationen und InteraktionenKreuzresistenz: Einige Mutationen, die unter einem bestimmten Medikament generiert werden, resultieren in einer verminderten Wirksamkeit oder sogar dem vollständigen Wirkungsverlust von mehreren oder allen Substanzen einer Klasse. Therapeutische Folgeoptionen können dadurch stark limitiert werden. Kompensatorische Mutationen: Oft vermindern Resistenz-assoziierte Mutationen die virale Fitneß. Das Virus versucht dies mit der Akkumulation von weiteren spezifischen Mutationen, die die Vermehrungsrate des Virus wieder erhöhen, zu kompensieren Interaktionen: Mutationen, die verläuft nicht über einen einheitlichen Resistenzpfad und ist dementsprechend genotypisch schwierig nachzuweisen das Virus für eine oder mehrere antiretrovirale Substanzen resistent machen, können dazu führen, daß andere Substanzen (wieder) sensitiver werden. Ein Beispiel ist die Mutation M184V die Resistenz gegen 3TC und FTC bewirkt, aber eine bestehende Resistenz gegen AZT minimieren oder je nach Resistenzgrad wieder aufheben kann. 6. Genetische Resistenzbarriere und antiretrovirale SubstanzenDie Resistenzentwicklung ist ein gradueller Prozess. Je nach antiretroviraler Substanz sind eine, wenige oder zahlreiche Mutationen zum Erreichen einer starken Resistenz notwendig. Unter genetischer Barriere versteht man die Anzahl an Mutationen die zur Resistenzbildung in Abhängigkeit von der Zeit notwendig sind. Geboostete Protease Inhibitoren besitzen die höchste genetische Barriere. Unter einer Ersttherapie mit Protease Inhibitoren entstehen bei virologischem Versagen äusserst selten Resistenz-assoziierte Protease Mutationen. Durch den 'Booster-Effekt' mit Ritonavir oder Cobicistat wird die Resistenzbildung bei Protease Inhibitoren wahrscheinlich erschwert. Für die Medikamente der anderen Substanzklasse reichen meist ein bis zwei Mutationen zur vollständigen Resistenzbildung aus. Insbesondere bei den NNRTI zeigte sich mit der neuen Substanz Rilpivirin, dass die Höhe der Ausgangsviruslast ein entscheidender Faktor für die Resistenzbildung darstellt. Entsprechend der Zulassungskriterien darf Rilpivirin somit nur bei Patienten mit niedrigen Viruslastwerten von weniger als 100.000 HIV-1 RNA Kopien/ml zur Vermeidung einer Resistenzentwicklung eingesetzt werden. Bei der neuen Substanzklasse der Integrase Inhibitoren reichen zum Wirkverlust ein bis zwei spezifische Resistenzmutationen. Dolutegravir scheint eine hohe Resistenzbarriere zu haben und zeigt oftmals noch eine Wirksamkeit gegen Viren mit spezifischen Resistenzmutationen im Integrasegen. Bei den Fusions Inhibitoren ist eine Resistenzmutation oftmals für ein virologisches Versagen ausreichend. Die Datenlage zur Resistenzbildung bei den CCR5 Antagonisten verläuft nicht über einen einheitlichen Resistenzpfad und ist dementsprechend genotypisch schwierig nachzuweisen (s. Kap. 7). Jedoch ist die genetische Barriere alleine nicht für einen Therapieerfolg aussagekräftig, wie anhand des Beispiels von Retrovir, welches eine höhere genetische Barriere als Abacavir oder Tenofovir besitzt, verdeutlicht wird. So war Retrovir im Rahmen von klinischen Studien dem Abacavir oder Tenofovir unterlegen. Vertäglichkeit und Potenz sind zwei weitere Parameter, die zum Therapieerfolg beitragen. Weiterhin soll im klinischen Alltag die Einzelsubstanz nicht alleine betrachtet, sondern immer unter Berücksichtigung der Kombinationspartner beurteilt werden. 7. Tropismusbestimmung und Resistenzentstehung unter CCR5-AntagonistenCCR5-Antagonisten, wie Maraviroc oder Vicriviroc, sind nur wirksam, wenn CCR5-trope HI-Viren vorliegen und keine CXCR4-tropen beziehungsweise dual-tropen Viren nachweisbar sind. Somit muss vor dem Einsatz von CCR5-Antagonisten ein Tropismustest durchgeführt werden. In Analogie zur Resistenzanalyse kann die Tropismusbestimmung genotypisch oder phänotypisch durchgeführt werden. Ein wesentlicher Vorteil des kostengünstigeren genotypischen Verfahrens ist die schnelle Ergebnismitteilung. In Akutsituationen kann ein Ergebnis binnen weniger Arbeitstage vorliegen. Die Interpretation der genotypischen Tropismusbestimmung erfolgt mittels einer bioinformatischen Analyse (http://www.geno2pheno.org). Mittels der genotypischen Analyse ist eine Tropismusbestimmung bei nicht nachweisbarer Viruslast aus proviraler DNA durchführbar. Die Übereinstimmung zwischen genotypischen (geno2pheno) und phänotypischen Testergebnissen (Standard Trofile) beträgt ca. 85%. In Deutschland wird nur noch der genotypische Test durchgeführt und vergütet. Das Ergebnis wird mittels des freiverfügbaren Interpretationssystems geno2pheno generiert. Das mit dem System ermittelte Ergebnis ist die "Falsch-Positivrate" (FPR). FPR von >15% bedeutet, dass R5-trope Viren vorliegen und Werte von <5% werden mit X4-tropen Viren assoziiert. Dazwischen liegt der Graubereich in dem eine Aktivität diskutierbar ist. Mittels einer erneuten Tropismusanalyse kann überprüft werden, ob ein Therapieversagen durch die Selektion von CXCR4- bzw. Misch- oder dual-tropen HI-Viren beruht. Falls dies der Fall ist, ist ein erneuter Einsatz des gleichen oder eines anderen CCR5-Antagonisten nicht mehr sinnvoll. Therapieversagen mit CCR5 Anatagonisten kann sowohl durch Tropismuswechsel als auch durch Resistenzentwicklung bedingt. Durch die genotypische Analyse des V3 Bereiches und anderer Bereiche des gp120 Gens kann man untersuchen, ob das Versagen bei Vorliegen von R5-tropen Viren durch die Entstehung von resistenten HIV-Varianten zu erklären ist. Denn ein CCR5-tropes Virus kann der Inhibition durch einen CCR5-Antagonisten durch Mutationen innerhalb des env-Gens ausweichen. Hierbei wird es dem Virus ermöglicht, den CCR5-Rezeptor zu nutzen, obwohl ein CCR5-Antagonist daran gebunden ist. Es wurden bisher keine typischen Muster zur Resistenzentstehung identifiziert. 8. Deutsch-Österreichische Leitlinien (DAIG 2012)
|
||||||||||||||||||||||||||||||||||||||||||||||||||||




Seite zuletzt geändert am 26.08.2015 08:50:00 |